责编 | 酶美
来源 | BioArt
在动物体内特定的细胞群体中进行时空特异性的细胞标记和基因操作一直是生物学研究的技术难题。位点特异性重组酶,尤其是基于Cre重组酶而开发的各种诱导型重组酶系统被广泛用于细胞标记和基因功能研究【1】。其中光诱导的Cre重组酶系统不仅安全、易调节且具有高度的时空分辨率【2,3】。然而单个重组酶系统具有一定的局限性,很难用于解析复杂的细胞网络,尤其是神经系统。Dre重组酶来源于D6噬菌体,能特异性地识别一段长度为32bp的DNA序列(rox位点)并介导该序列间的DNA发生重组【4】。因其重组效率高且不会与Cre重组酶发生交叉反应,二者常联合使用以实现更加精细的基因操作【5】。与Cre重组酶一样,研究者也相继开发出多种诱导型Dre重组酶,但均为化学诱导系统6。其诱导周期长、效率低且缺乏空间特异性。这些缺点都极大地限制了重组酶在特定细胞类群中实现精准的基因重组。
2020年12月14日,华东师范大学李大力与阳怀宇、刘明耀课题组合作,在PNAS杂志上发表了题为Efficient photoactivatable Dre recombinase for cell type-specific spatiotemporal control genome engineering in the mouse的研究论文。该研究将蛋白质动态构象理论计算与光遗传学相结合,开发出了高效、严格受蓝光调控的光激活Dre重组酶系统(photoactivatable Dre, PA-Dre)并结合组织特异性Cre构建了由蓝光和Cre双调控的新型光诱导Dre重组酶系统,命名为Cre激活的光诱导Dre(Cre-Activated Light Inducible Dre, CALID)系统,从而在特定细胞类群中实现了高效的时空特异性基因重组和细胞标记,为体内基因操作增加了更为精准可控的新工具。

为了构建出光响应速度快且组份简单的新型Dre重组酶系统,该研究采取了光诱导分段蛋白重组策略【7】。由于目前还没有报道过Dre重组酶的晶体结构,如何找到有效的拆分位点成为一个难点。借助阳怀宇课题组动态构象研究技术,团队通过计算,预测了将完整的Dre重组酶拆分成两个片段的一系列拆分位点。通过组合筛选,确认分别将Dre重组酶的N端(第1-246个氨基酸)和C端(第247-343个氨基酸)与蓝光调控的Magnets系统【8】融合表达时能更好地响应蓝光,从而高效、严谨地诱导基因重组(图1)。
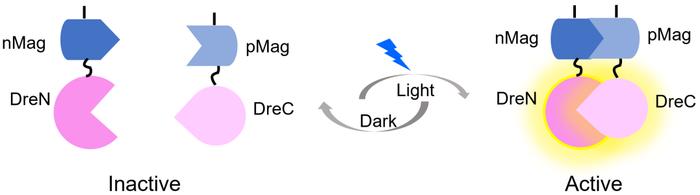
图1. PA-Dre系统的工作原理。在没有蓝光的情况下,分段的Dre重组酶因相距较远,没有催化活性。接受蓝光刺激后,分段的Dre重组酶在光敏蛋白的带动下快速二聚化形成具有完整功能的Dre重组酶即可催化重组反应。
为了降低本底同时增加光诱导Dre重组酶的诱导效率,研究者针对各元件的相对位置、连接氨基酸序列、核定位信号序列的种类及位置进行了一系列优化,最终得到了诱导倍数高达100倍的PA-Dre系统。在HEK-293T等细胞上测试结果表明该系统具有良好的光照时间和光照强度依赖性及高度的时空特异性(图2)。
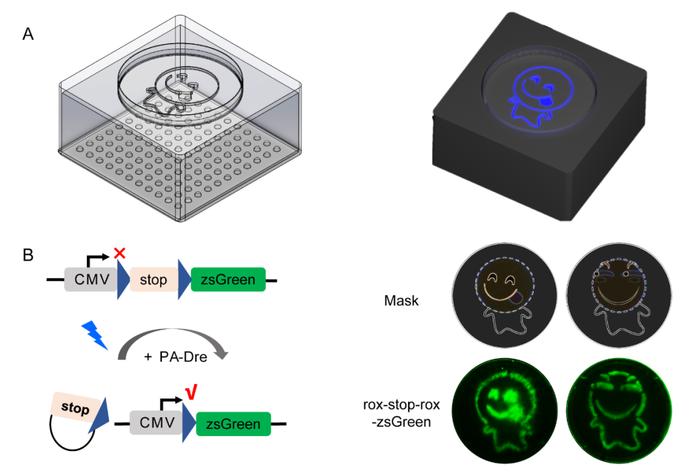
图2. PA-Dre系统的空间特异性(A)用于证明 PA-Dre 空间特异性的照光装置示意图。左侧为透视图,右侧为实物图。在黑色的盒子底部有一排蓝色的 LED,顶部有一个9厘米的圆孔,上边放置了一个没有盖子的培养皿,其底部覆盖了黑色镂空图案。(B)PA-Dre 和 rox-stop-rox- zsGreen 共同转染 HEK-293T细胞,将 10 cm皿放置在底部覆盖黑色镂空图案的装置上进行光照处理。蓝色的光透过小缝隙照射到对应区域的 HEK-293T 细胞,激活该区域的 PA-Dre 发生重组反应,切掉转录终止元件引起 zsGreen 的表达(左图),最终形成了可爱的表情包图案(右图,上排为黑色镂空图案,下排为zsGreen表达后形成的图案)。
接下来,研究者分别利用流体动力学尾静脉注射质粒及腺相关病毒的方法探究了PA-Dre系统在小鼠体内的作用效果。研究结果表明该系统不仅可以激活外源基因表达,还可在小鼠的肝脏和脑部高效地调控内源基因表达。通过控制注射质粒的量、病毒滴度及光照参数可以灵活地调节被标记细胞的区域和数目,但由于启动子的限制,还不能实现特定细胞类群的时空特异性基因重组。
为了实现更高分辨率的基因操作并扩大PA-Dre重组酶系统的适用性,该研究基于双重倒置开放阅读框策略构建了Cre激活的光诱导Dre(CALID)系统(图3),并在小鼠大脑中对其性能进行了测试。分别借助腺相关病毒表达的CaMKIIα-EGFP-2A-Cre病毒和PV-Cre转基因小鼠,调控光诱导的Dre重组酶,通过调节光照参数和光纤直径大小,实现了对CaMKIIα兴奋性神经元和Parvalbumin抑制性神经元的密集和稀疏型标记。与传统的稀疏细胞标记方法【9】相比,CALID介导的细胞标记更加高效灵活,仅改变光照参数即可实现不同区域和强度的酶活调节和细胞标记。
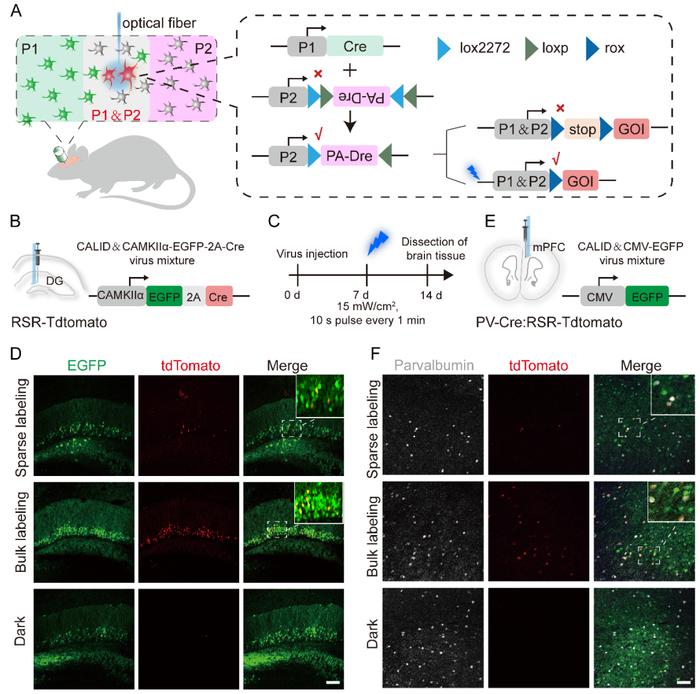
图3. 利用 CALID 系统时空特异性地标记特异性神经元细胞。(A) P1和P2分别是Cre和CALID的特异性启动子,P1&P2是既表达Cre又表达CALID的区域,在该区域Cre能够修复PA-Dre 编码框的方向使其正常表达,在接受蓝光刺激后发挥功能。(B-D) 在R26-RSR-tdTomato小鼠海马DG区注射CALID和CaMKIIα-EGFP-2A-Cre病毒标记CaMKIIα兴奋性神经元。(E-F) 在PV-Cre:R26-RSR-tdTomato小鼠大脑皮层注射CALID和CMV-EGFP病毒标记PV抑制性神经元。
综上所述,本研究开发了蓝光激活的Dre重组酶系统并在此基础上构建了Cre激活的光诱导Dre系统,进一步提高了基因重组工具的精准度。与已有的光诱导重组酶系统相比,CALID系统具有更强的普适性,可作为通用型工具与特异性Cre重组酶联合使用,从而在特定类型的细胞中实现时空特异性的基因重组。今后计划构建CALID系统定点整合的转基因小鼠,这样能更好地与组织特异性的Cre重组酶的转基因小鼠资源库相结合,为精确的标记细胞和调控基因的表达提供体内研究的新工具。
据悉,华东师范大学博士研究生李慧莹、张乾森副研究员和顾怡然副研究员为本文共同第一作者,刘明耀教授、阳怀宇研究员、李大力研究员为本文共同通讯作者,中科院周斌研究员,华东师范大学叶海峰和林龙年研究员对本项研究提供了大力支持。
原文链接:
https://www.pnas.org/content/early/2020/12/10/2003991117
排版人:SY
参考文献
向上滑动阅览
1. Meinke, G., Bohm, A., Hauber, J., Pisabarro, M. T. & Buchholz, F. Cre Recombinase and Other Tyrosine Recombinases. Chem. Rev. 116, 12785–12820 (2016).
2. Kawano, F., Okazaki, R., Yazawa, M. & Sato, M. A photoactivatable Cre–loxP recombination system for optogenetic genome engineering. Nat. Chem. Biol. 12, 1059–1064 (2016).
3. Taslimi, A. et al. Optimized second-generation CRY2-CIB dimerizers and photoactivatable Cre recombinase. Nat. Chem. Biol. 12, 425–430 (2016).
4. Sauer, B. & McDermott, J. DNA recombination with a heterospecific Cre homolog identified from comparison of the pac-c1 regions of P1-related phages. Nucleic Acids Res. 32, 6086–6095 (2004).
5. He, L. et al. Enhancing the precision of genetic lineage tracing using dual recombinases. Nat. Med. 23, 1488–1498 (2017).
6. Anastassiadis, K. et al. Dre recombinase, like Cre, is a highly efficient site-specific recombinase in E. coli, mammalian cells and mice. DMM Dis. Model. Mech. 2, 508–515 (2009).
7. Dagliyan, O. & Hahn, K. M. ScienceDirect Controlling protein conformation with light. Curr. Opin. Struct. Biol. 57, 17–22 (2019).
8. Kawano, F., Suzuki, H., Furuya, A. & Sato, M. Engineered pairs of distinct photoswitches for optogenetic control of cellular proteins. Nat. Commun. 6, 1–8 (2015).
9. Lin, R. et al. Cell-type-specific and projection-specific brain-wide reconstruction of single neurons. Nat. Methods 15, 1033–1036 (2018).